% \ VignetteIndexEntry{1。界面教程}%\VignetteKeywords{用户界面}%\VignettePackage{BioNetStat} %\VignetteEngine{knitr::knitr}
从细胞和细胞器到整个生物圈的生物系统中发生的相互作用的多样性,可以用网络理论的工具进行评估。结构的动态性和要素之间相互作用的动态性是这些系统的固有特征。已经提出了几种工具来比较网络,代表一个系统假设的许多状态。然而,到目前为止,还没有人能够同时比较两个以上网络的结构特征。由于一个给定的生物系统可以假设许多状态,我们开发了一种统计工具来比较两个或多个网络,并指出系统中的关键变量。考虑到之前的数据预处理,BioNetStat能够比较由基因表达数据、代谢物浓度或任何可能具有统计相关性的变量构建的相关网络。网络比较是基于图谱(邻接矩阵中的特征值组),如谱分布。这一测量与网络的几个结构特征有关,如行走的数量、直径和点击。除了光谱分布,BioNetStat还可以使用光谱熵、度分布和节点中心来比较网络。到目前为止,BioNetStat的理论基础仅在硕士论文,用葡萄牙语。这篇论文正在制作中,将尽快在这里发表。
安装R和BioNetStat,如图所示自述,您只需打开R并运行以下命令。
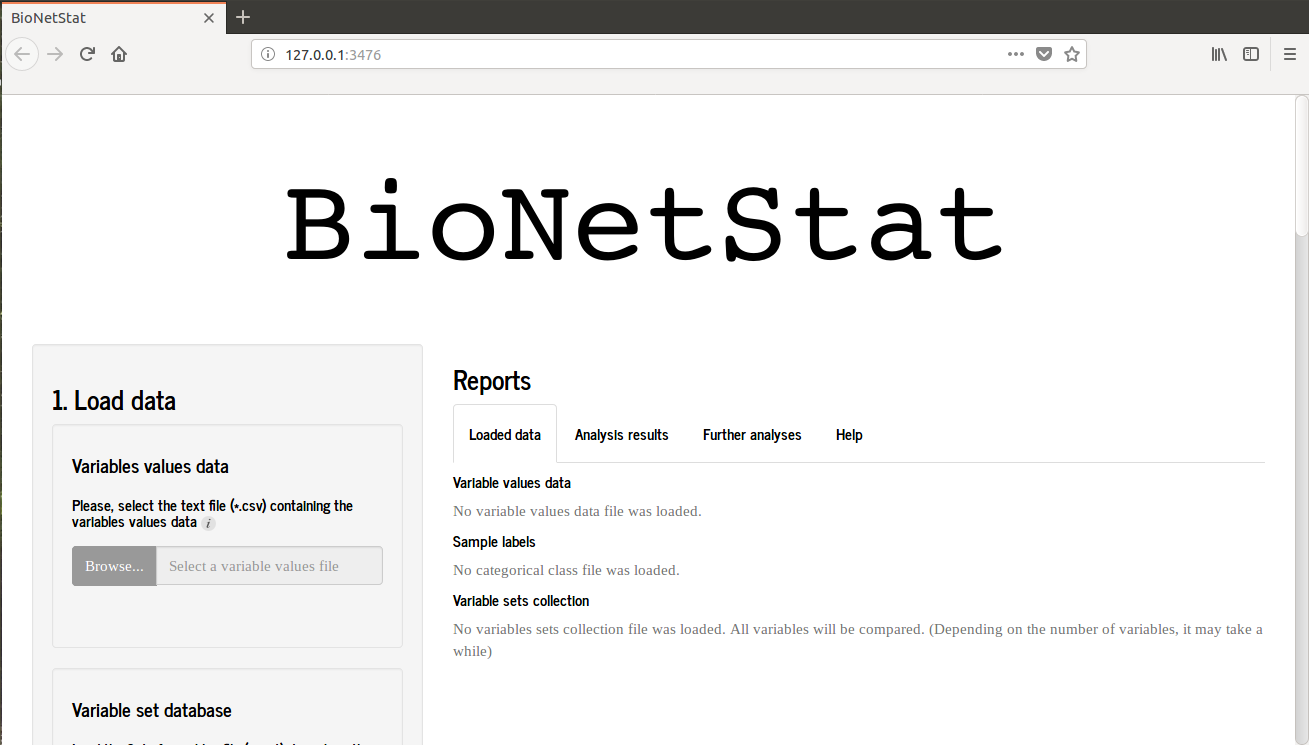
runBioNetStat()当你启动BioNetStat时,你会看到这张图片。

如果您正在使用Rstudio将打开一个替代窗口,因此BNS将在浏览器中使用时表现良好。所以,点击屏幕左上方的“在浏览器中打开”,BNS将作为第一张图片在浏览器中打开。

点击“浏览..”'图标在第1节(加载数据)将打开一个窗口的变量值表被选择。

在本教程中,要选择的文件是variablesValue_BioNetStat_tutorial_data.csv.文件将被加载,软件将识别由r分类为“数字”的列,屏幕上显示了表的前一个视图。此外,“因素”部分将识别哪些列被r评为“因素”。在这个部分中,您可以选择将用于选择比较的状态(处理,条件)的因素,每个状态将是一个要比较的网络。状态的选择可以在“Choose The conditions to be comparison”中完成:

选择变量值表后,可以将文件“变量组”(可选)加载到“变量集数据库”中。在本教程中,所选文件为variableSet_BioNetStat_tutorial_data.gmt根据相关的代谢途径来设定变量组。如果用户没有加载任何文件,程序将比较网络与文件“变量值数据”中加载的所有变量


构建变量组表的教程如下在这里.
加载输入文件后,可以将参数设置为网络构造:将在“变量集大小范围”中比较的网络中的变量(节点)数量的低和高。程序返回介于这些值之间的变量集的数量(上图)。2.相关性度量用于推断相关网络。3.关联强度将被用作两个节点之间形成边的阈值,以及使用哪个阈值。4.要构建的网络类型,在边缘上有或没有权重。在有权重的网的情况下,哪个关联度量将被用作边权重。




加载文件并选择分析参数后,单击“开始分析”来比较网络。







您需要插入一个包含两列的表,其中第一列必须是您正在研究的变量的名称,第二列是这些变量各自的KEGG代码。KEGG代码可以在KEGG的的网站。如果研究的变量是基因/蛋白质或代谢物,除了表中变量的代码外,还应选择在绘图中使用的颜色。图中出现的变量与节点微分分析表中分析的变量相同,可以根据test的值或与其关联的pvalor或qvalue进行过滤。有必要选择哪一个代谢途径将用于产生可视化和在哪个物种.

在KEGG地图上,你会注意到矩形或圆形被分为列,代表你选择比较的治疗方法。颜色的强度是相对于根据图例选择的中心值。这是一个用kegg的癌症(05200)途径的基因构建的人类(“hsa”)的地图示例。

当点击保存按钮时需要等待程序下载地图,将其保存在“下载”目录下的压缩文件中。视图位于压缩文件中。

可视化网络可以帮助我们更容易地理解变化的模式。这种可视化可以通过几种方式实现,其中一些可以在BioNetStat的“网络可视化图”选项卡中使用。由于本节中的BNS可以比较多个状态,每次只需要选择其中两个进行查看和比较。






BioNetStat还提供了对变量行为的研究,以便用户可以对其研究对象有一个完整的视图。1.最初,可以通过构造热图来探索变量,在热图中选择图形的参数。列中是样本,颜色表示每个样本属于哪个状态。在“热图集群选项”一节中,可以对热图的列(样本)和行(变量)进行分组。



在KEGG地图上,你会注意到矩形或圆形被分为列,代表你选择比较的治疗方法。颜色的强度是相对于根据图例选择的中心值。这是一个用kegg的癌症(05200)途径的基因构建的人类(“hsa”)的地图示例。

当点击保存按钮时需要等待程序下载地图,将其保存在“下载”目录下的压缩文件中。视图位于压缩文件中。


